Vilniaus universiteto (VU) Gyvybės mokslų centro (GMC) tyrėjų komanda atrado unikalų būdą, kaip ląstelės gali nutildyti tam tikrus genus nekirpdamos DNR. Šis novatoriškas tyrimas, vadovaujamas dr. Patricko Pauscho, buvo paskelbtas žurnale „Nature Communications“. Jo naujumas – genų nutildymas, tarsi paspaudžiant „pauzės“ mygtuką tam tikroms genetinėms instrukcijoms ląstelėse.
Doktorantė Rimvydė Čepaitė, dr. Aistė Skorupskaitė, studentė Gintarė Žvejytė ir dr. P. Pauschas, dirbdami kartu su tarptautine mokslininkų komanda, atrado specifinę sistemą, gebančią ląstelėje surasti ir nutildyti nepageidaujamą DNR. Ši sistema gali prisidėti kuriant saugesnius genų modifikavimo metodus, o ateityje padėti gydyti genetinių klaidų sukeliamas ligas.
„Skirtingai nei gerai žinoma CRISPR genų redagavimo sistema, dažnai apibūdinama kaip „molekulinės žirklės“, naujai ištirta IV-A tipo CRISPR sistema nekerpa genų. Užuot tai dariusi, ji naudoja į DNR nukreiptą „efektoriaus“ kompleksą, įdarbinantį fermentą DinG, kuris judėdamas DNR grandine geba subtiliai nutildyti tikslinius genus“, – aiškina dr. P. Pauschas.
Pasak mokslininko, įdomu, kaip ši sistema sugeba atpažinti tikslią DNR vietą, kur ir pradeda veikti: „Sistema naudoja du baltymus (Cas8 ir Cas5), kad surastų labai trumpą sekos motyvą, esantį šalia taikininės DNR sekos. Kai abu baltymai atpažįsta šią trumpą seką, jie atskiria dvi DNR grandines, kad galėtų atlikti taikininės sekos patikrą.“
Svarbus šio proceso etapas – R-kilpų (R-loop) susidarymas. Tai – atviros DNR struktūros, kuriose gidinė RNR susijungia su savo DNR taikiniu taip signalizuodama sistemai, kad gali būti pradėtas genų nutildymo procesas.
„Raidė „R“ R-kilpoje žymi RNR. Visos su DNR sąveikaujančios CRISPR-Cas sistemos naudoja šią struktūrą, kad patikrintų DNR seką ir surastų teisingą taikinio vietą. Stabilios R-kilpos formuojasi tik tada, kai DNR seka pakankamai atitinka gidinę RNR. R-kilpa iš esmės signalizuoja sistemai, kada pradėti geno nutildymą“, – teigia VU GMC mokslininkas.
Pasak jo, DinG fermentas dar labiau sustiprina genų slopinimą atsukdamas DNR grandines. Todėl sistema geba paveikti ilgesnę DNR seką.
Šis atradimas leis ateityje redaguoti genomus nekerpant DNR ir kurti moksliniuose tyrimuose bei biotechnologijose tiksliau veikiančius įrankius.
„Unikalus mūsų sukurtos sistemos gebėjimas pereiti per DNR nekarpant genų atveria naujas galimybes pažangioms genų redagavimo technologijoms. Ši nauja metodika gali turėti teigiamą poveikį visuomenei, leisdama saugiau modifikuoti genus“, – sako dr. P. Pauschas.
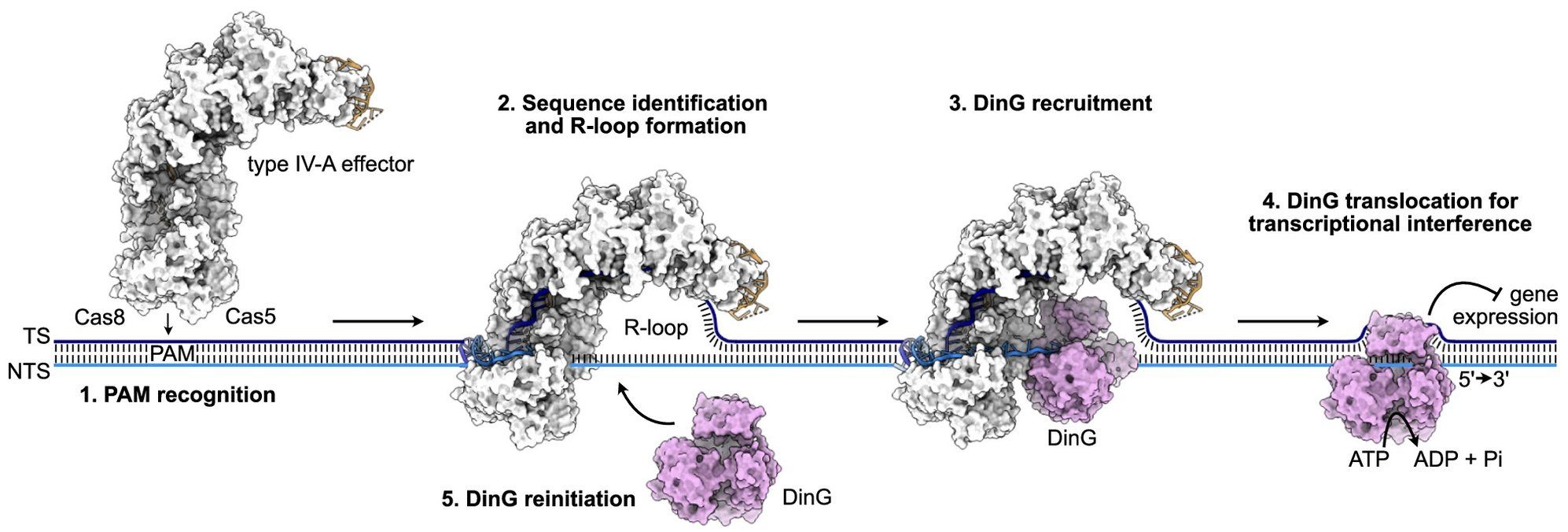
Šioje iliustracijoje pavaizduotas galutinis IV-A tipo CRISPR mechanizmo modelis. Iš kairės: 1 žingsnis rodo „efektoriaus“ komplekso, sudaryto iš gidinės RNR ir IV-A tipo baltymų, susidarymą (atvaizduota iš krioEM struktūros) ir kaip šis kompleksas prisijungia prie trumpo motyvo, esančio šalia tikslinės DNR vietos (PAM). 2 žingsnyje susidaro R-kilpa, kuri signalizuoja, kad rasta taikinio vieta DNR grandinėje. 3 žingsnyje matomas DinG prisijungimas (rožinės spalvos burbulo formos struktūra). 4 žingsnyje DinG greičiausiai juda išilgai DNR, kad nutildytų geną, ir gali pakartoti procesą toliau nutildydama geną (5 žingsnis).
